Coronaviren
Coronaviren sind verantwortlich für eine Reihe von Erkrankungen bei Menschen, Säugetieren (außer Affen) und Vögeln. Besonders häufig findet man Coronaviren in Fledermausarten auf der ganzen Welt. Sie verursachen unter anderem Atemwegs- und Magen-Darm-Erkrankungen. Beim Menschen sind sie die zweithäufigtse Ursache für Erkältungskrankheiten.
Das erste Coronavirus wurde in den 1930er Jahren entdeckt und beschrieben und war Auslöser einer infektiösen Bronchitis in Hühnern, das avian infectious bronchitis virus (IBV). Erst 1965 wurde das erste humanpathogene Coronavirus entdeckt; kurz danach dann ein weiteres, heute bekannt als HCoV-229E, HCoV-OC43 [1,2]. Erst 2003 wurde mit SARS-CoV ein weiteres humanpathogenes Coronavirus gefunden. In der Folge entdeckte und charakterisierte man noch weitere: MERS-CoV (2012), HCoV-NL63 (2004), HCoV-HKU1 (2005), SARS-CoV-2 (2020). Damit sind bis heute, Februar 2023, sieben humanpathogene Coronaviren bekannt. In Fledermäusen fand man außerdem etliche weitere die prinzipiell in der Lage sind auf den Menschen überzuwechseln.
 Aus: Bauernfeind et al, Zoonoses: Infectious Diseases Transmissible Between Animals and Humans,ASM Press (2015)
Aus: Bauernfeind et al, Zoonoses: Infectious Diseases Transmissible Between Animals and Humans,ASM Press (2015)
Coronaviren werden der Ordnung Nidovirales zugeordnet und bilden dort eine eigene Virenfamilie, Coronaviridae. In dieser Familie unterscheidet man vier Gattungen, Alpha-, Beta-, Gamma- und Delta-Coronavirus (Beispiel: SARS-CoV). Angehörige der Gattungen Alpha- und Betacoronavirus infizieren hauptsächlich Säugetiere, Gammacoronaviren wurden bei Vögeln gefunden.
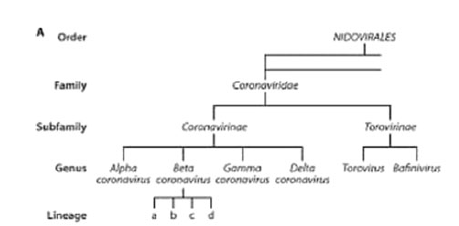
Aus: Jaiswal, N.K., Saxena, S.K. (2020). Classical Coronaviruses. In: Saxena, S. (eds) Coronavirus Disease 2019 (COVID-19). Medical Virology: From Pathogenesis to Disease Control. Springer, Singapore.
Coronaviren sind rund und haben einen Durchmesser von etwa 50 bis 200 nm. Sie sind von einer Hülle aus Lipiden in die verschiedene Proteine eingelagert sind umgeben. Diese Proteine geben den Virionen ihr charakteristiches Aussehen.
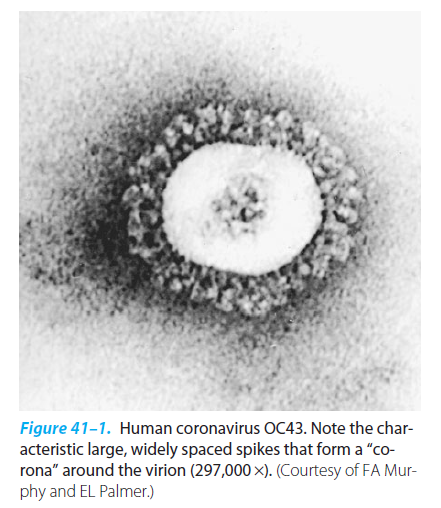
Aus: Brooks, Medical Microbiology, 24th edition, McGraw-Hill (2007), S.568
Das Genom, mit 27 KB bis 32 KB das größte RNA-Genom in Viren, besteht aus einem einzeln Strang RNA und enthält die Anleitung zur Herstellung der verschiedenen Proteine die zur Vervielfältigung der RNA und zur Bildung neuer Viruspartikel notwendig sind. Diese sogenannte (+)ssRNA kann direkt als Vorlage von den Ribosomen der Wirtszelle verarbeitet werden.
Nachdem das Virus in die Zelle eingedrungen dient die jetzt freiliegende RNA als Vorlage für die Produktion einer sogenannten RNA-Polymerase. Diese fertigt Gegengleiche Kopien der Viren-RNA an die dann entweder als Vorlage für die mNA der verschiedenen Proteine als auch für neue Viren-RNA dient. Insgesamt findet man auf dem Genom die Bauanleitung für etwa 20 Proteine mit verschiedenen Aufgaben. Sobald die für neue Viruspartikel notwendigen Bestandteile hergestellt wurden werden sie verpack und treten aus der Zelle aus. Dabei erhalten sie ihre, mit verschiedenen Proteinen dekorierte, Lipidhülle. (für eine detaillierte Beschreibung dieses komplexen Prozesses siehe die angegebene Literatur)
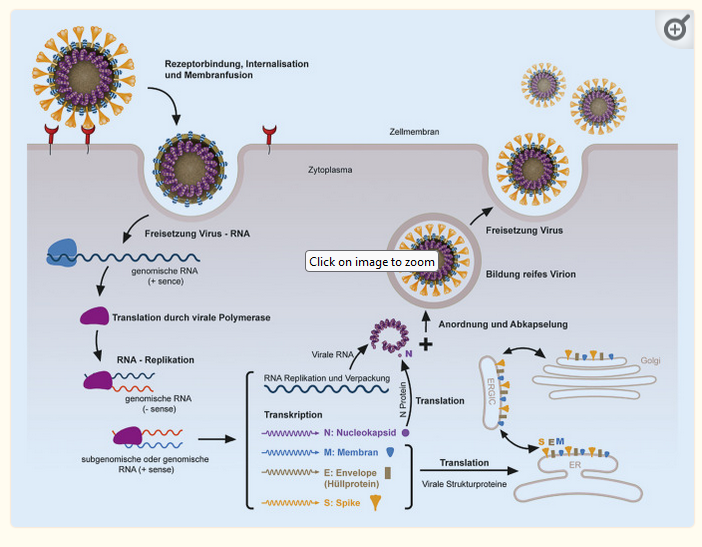
Quelle: Grundlagen der Replikation und der Immunologie von SARS-CoV-2
Prominente Vertreter sind die Erreger von SARS, MERS und Covid-19. Alle drei verursachen schwerwiegende Erkrankungen.
SARS (severe acute respiratory syndrome) trat 2002 zuerst in China auf und verbreitete sich von dort in 32 Länder. Das Virus SARS-CoV war verantwortlich für eine schwere Erkrankung der Atemwege und forderte im Verlauf der Pandemie mit etwa 8100 Erkrankten weltweit 774 Todesfälle. Das entspricht einer Mortalität von knapp 10%. Die Ausbreitung endete 2003/2004 abrupt und seitdem sind keine neuen Fälle aufgetreten. (SARS (Schweres Akutes Respiratorisches Syndrom))
2012 wurde bei einer Person aus dem arabischen Raum eine Erkrankung mit ähnlichen Symptomen festgestellt. Als Erreger wurde ebenfalls ein Coronavirus, MERS-CoV, identifiziert. MERS (Middle East respiratory syndrome) tritt überwiegend auf der arabischen Halbinsel auf und wird von Dromedaren auf den Menschen übertragen. Bis heute, Februar 2023, sind mehr als 2400 Fälle in 27 Ländern bekannt; mehr als 800 Menschen starben. Das entspricht einer Mortalität von über 30%. (Informationen des RKI zu MERS-Coronavirus
Für beide Erkrankungen gibt es weder spezifische Medikamente noch zugelassene Impfstoffe.
Im Dezember 2019 traten in China wieder schwere Lungenentzündungen unbekannter Ursache auf. Wenige Wochen später hatte man den Erreger isoliert und sequenziert. Aufgrund seiner Ähnlichkeit mit SARS-CoV nannte man das neu aufgetauchte Virus SARS-CoV-2. Die Erkrankung bekam den Namen Covid-19 (Coronavirus disease 2019). Das Virus verbreitete sich innerhalb weniger Monate auf der ganzen Welt. Bis heute (10.Februar 2023) gab es 755.385.709 bestätigte Erkrankungs- und 6.833.388 Todesfälle. Die zur Eindämmung notwendigen Maßnahmen wie Pflicht zum Tragen von medizinischen u/o FFP2-Masken, Kontaktbeschränkungen usw konnte eine Überlastung der Gesundheitssysteme zumindest zum Teil verhindert werden. Seit Dezember 2020 existieren wirksame und gut verträgliche Impfstoffe die, z.T. aufbauend auf den Forschungen an SARS und MERS, in Rekordzeit entwickelt werden konnten und die Schwere der Erkrankung reduzieren. Inzwischen gibt es auch spezifische Medikamente die man vor allem bei besonders gefährdeten Personengruppen einsetzt. (COVID-19 (Coronavirus SARS-CoV-2))
Inzwischen sind in den meisten Ländern die Maßnahmen zur Eindämmung der Pandemie wieder weitgehend aufgehoben worden und man kehrt zu der normalen Lebensweise vor Covid zurück. Die wirtschaftlichen und gesellschaftlichen Folgen werden uns alle aber sicher noch eine Weile beschäftigen.
Bis heute ist nicht klar woher die Viren ursprünglich kamen und auf welchen Wegen sie auf den Menschen übergesprungen sind. Die Geschichte der letzten drei Jahre sollte uns aber bewusst machen dass so etwas jederzeit wieder passieren kann.
Literatur:
- Tyrrell DA, Bynoe ML. Cultivation of viruses from a high proportion of patients with colds. Lancet. 1966 Jan 8;1(7428):76-7. https://doi.org/10.1016/s0140-6736(66)92364-6
- Hamre D, Procknow JJ. A new virus isolated from the human respiratory tract. Proc Soc Exp Biol Med. 1966 Jan;121(1):190-3. https://doi.org/10.3181/00379727-121-30734
- Hu, B., Ge, X., Wang, LF. et al. Bat origin of human coronaviruses. Virol J 12, 221 (2015).
https://doi.org/10.1186/s12985-015-0422-1 - Banerjee, A.; Kulcsar, K.; Misra, V.; Frieman, M.; Mossman, K. Bats and Coronaviruses. Viruses 2019, 11, 41.
https://doi.org/10.3390/v11010041 - Jaiswal, N.K., Saxena, S.K. (2020). Classical Coronaviruses. In: Saxena, S. (eds) Coronavirus Disease 2019 (COVID-19). Medical Virology: From Pathogenesis to Disease Control. Springer, Singapore.
https://doi.org/10.1007/978-981-15-4814-7_12 - V’kovski, P., Kratzel, A., Steiner, S. et al. Coronavirus biology and replication: implications for SARS-CoV-2. Nat Rev Microbiol 19, 155–170 (2021). https://doi.org/10.1038/s41579-020-00468-6
- Woo, P. C., Lau, S. K., Huang, Y., & Yuen, K. Y. (2009). Coronavirus diversity, phylogeny and interspecies jumping. Experimental biology and medicine (Maywood, N.J.), 234(10), 1117–1127. https://doi.org/10.3181/0903-MR-94
- Su, S., Wong, G., Shi, W., Liu, J., Lai, A. C. K., Zhou, J., Liu, W., Bi, Y., & Gao, G. F. (2016). Epidemiology, Genetic Recombination, and Pathogenesis of Coronaviruses. Trends in microbiology, 24(6), 490–502.
https://doi.org/10.1016/j.tim.2016.03.003 - Corman, V. M., Muth, D., Niemeyer, D., & Drosten, C. (2018). Hosts and Sources of Endemic Human Coronaviruses. Advances in virus research, 100, 163–188. https://doi.org/10.1016/bs.aivir.2018.01.001
Weblinks:
Timeline of ECDC's response to COVID-19
WHO: Middle East respiratory syndrome coronavirus (MERS-CoV)
WHO: Coronavirus disease (COVID-19)
